Documentation :
Téléchargement :
Licence :
Qu’est ce que SIED ?
SIED est un logiciel gratuit et open-source d’analyse d’images isotopiques obtenues par microscopie SIMS (Spectrométrie de masse des ions secondaires).
Contexte scientifique :
SIED, Significant Isotopic Enrichment Detection est un outil conçu pour aider à l’analyse des images isotopiques SIMS (Secondary Ion Mass Spectrometry). Dans une telle analyse isotopique, au moins deux isotopes sont imagés, c’est à dire qu’une image représente la localisation d’un isotope et une autre image représente la localisation d’un autre isotope, les abondances relatives des deux isotopes étant supposées être constantes dans des échantillons « non enrichis ». Ceci peut être illustré par l’analyse de 12C14N et 12C15N pour lesquels l’abondance relative de 12C15N=12C15N/(12C14N+12C15N) est proche de 0,0037.
L’analyse SIMS conduit au comptage des ions secondaires, c’est à dire à une (petite) fraction des deux isotopes de l’échantillon, et accessoirement à des fluctuations statistiques dans la mesure des abondances isotopiques relatives. En fonction de la quantité totale d’ions secondaires détectés (les statistiques de comptage), les fluctuations statistiques induisent un « bruit » qui doit être comparé à la mesure. Plus les statistiques de comptages sont faibles, plus le rapport signal/bruit est faible et, incidemment, plus il est difficile de détecter les petits enrichissements en raison du marquage de l’échantillon dans l’un des deux isotopes.
Le but du logiciel SIED est de résoudre ce problème en comparant les abondances isotopiques mesurées aux fluctuations statistiques attendues (qui dépendent des statistiques de comptage) pour chaque pixel des images. Pour plus de détails, le principe des calculs de SIED a été publié dans (Delaune et al., 2013).
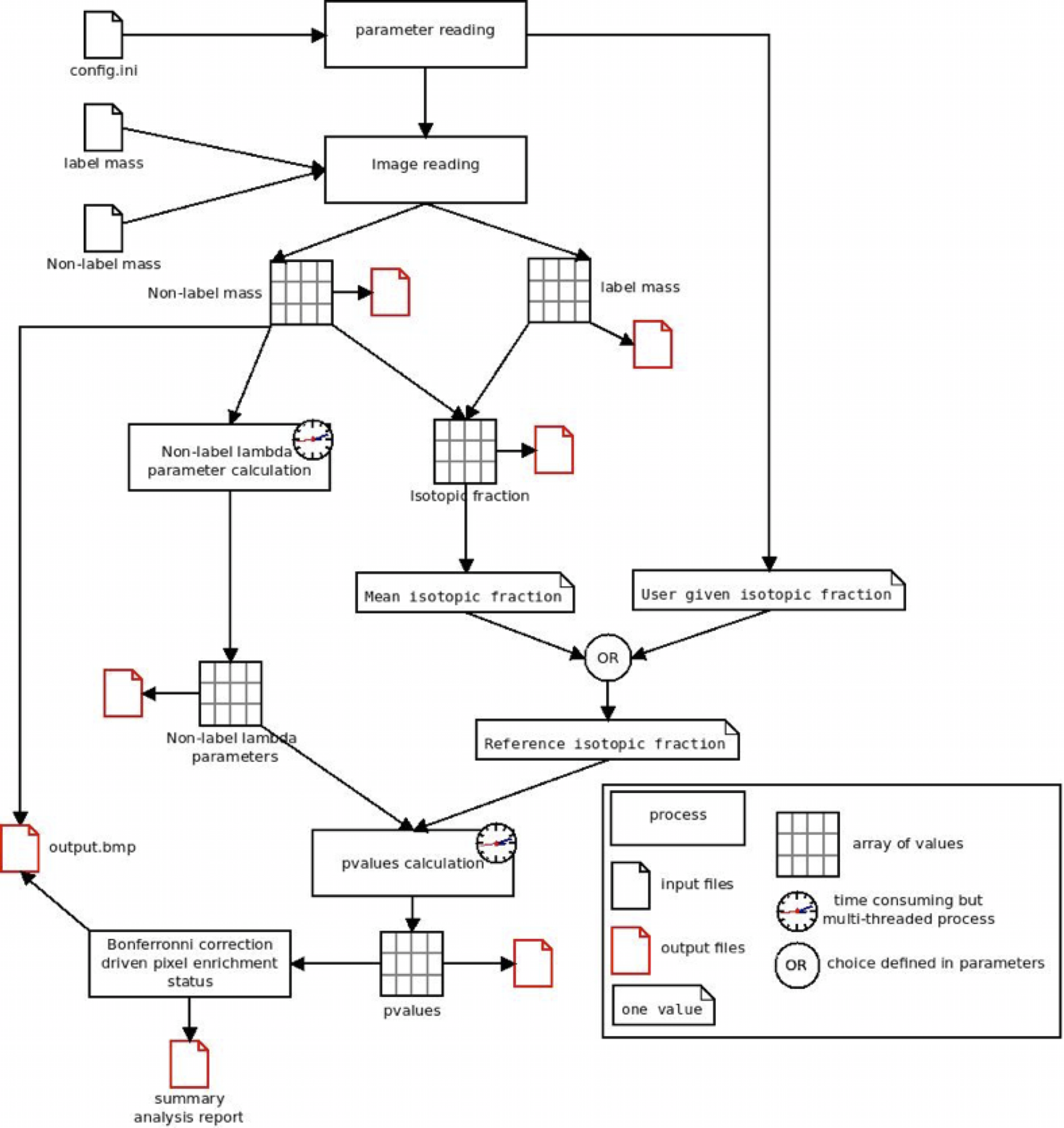
Brièvement, comme illustré, SIED ouvre le fichier de configuration (config.ini) qui contient les paramètres d’analyse. A partir de ces paramètres, SIED ouvre le fichier avec le nom correspondant à l’image de l’isotope désigné comme « label mass » (12C15N dans l’exemple précédent) et le fichier avec le nom correspondant à l’image de l’autre isotope désigné comme « non-label mass » (12C14N dans l’exemple précédent). Lorsque SIED ouvre ces fichiers, il convertit les images en tableaux où chaque cellule correspond à un pixel des images originales. SIED calcule ensuite la fraction isotopique pour chaque pixel (et crée un nouveau tableau), puis la fraction isotopique moyenne de l’image. Cette valeur peut être utilisée comme fraction isotopique de référence, c’est à dire la fraction isotopique considérée comme la valeur réelle de l’échantillon et à partir de laquelle des pixels de valeurs significativement différentes seront recherchés. Alternativement, la fraction isotopique de référence peut être entrée par l’utilisateur comme un paramètre. Pour estimer la distribution de la fraction isotopique mesurée autour de la fraction isotopique de référence, les statistiques de comptage sont estimées. A partir du tableau de masse non-marquée, SIED calcule le paramètre lambda de chaque pixel. Pour chaque pixel, la méthode consiste à rechercher la valeur du paramètre lambda qui maximise la somme des probabilités de Poisson (probabilité d’obtenir la valeur mesurée avec cette valeur de lambda) des pixels voisins. Le nombre de voisins retenus pour cette détermination est définie dans le fichier config.ini par le rang de la matrice, c’est à dire la distance (en pixels) autour du pixel analysé. En résumé, un rang 0 signifie que seul le pixel analysé est pris en compte, un rang 1 signifie que les 9 pixels du carré centré sur le pixel analysé sont pris en compte, un rang 2 signifie que les 25 pixels etc. L’effet quadratique du rang sur le nombre de calculs explique pourquoi cette étape prend beaucoup de temps. Ce temps peut être heureusement réduit par l’exécution parallèle de SIED (multi-thread). Une fois le tableau des paramètres lambda calculé, on calcule les p-value, c’est à dire (en termes simplifiés) la probabilité que la fraction isotopique mesurée ne diffère pas de la fraction isotopique de référence. Encore une fois, cette étape est longue mais accélérée par le multi-threading. Enfin, un test statistique est effectué (en utilisant la correction de Bonferronni pour les tests multiples) pour évaluer si chaque pixel est significatif ou non, et les fichiers de sortie sont sauvegardés.
Par extension, dans le cas d’échantillons enrichies isotopiquement dans lesquels une variation locale de l’enrichissement (enrichissement local supérieur ou inférieur par rapport à l’enrichissement moyen de l’échantillon) doit être analysée, un calcul similaire peut être effectué (test bilatéral).
Étape de préparation de l’analyse :
SIED nécessite que les images soient au format texte. Chaque valeur de pixel doit être un nombre entier positif (pas de séparateur décimal). Les pixels doivent être séparés par une tabulation.
En plus des images à analyser, SIED a besoin de paramètres pour l’analyse. Ces paramètres doivent être stockés dans un fichier (traditionnellement nommé config.ini) qui sera lu par SIED.
Les paramètres d’analyse peuvent être édités manuellement dans le fichier config.ini. Cependant, pour analyser une image NanoSIMS50, il est fortement recommandé d’utiliser SIEDmaker (pour le système Linux) ou SIEDmaker4win (pour le système Windows) pour générer ce fichier (voir SIEDusersguide pour plus d’informations).
Voir Installation sous Linux ou Installation sous Windows pour installer ces logiciels ainsi que SIED et son exécution.
Licence :
Ce programme est un logiciel libre : vous pouvez le redistribuer et/ou le modifier selon les termes de la Licence Publique Générale GNU telle que publiée par la Free Software Foundation, soit la version 3 de la Licence ou soit (à votre choix) toute version ultérieure.
Une copie du texte de la licence officielle (en anglais) est incluse dans le package à télécharger.
Contact :
Pou plus d’informations ou pour toutes questions, veuillez contacter :
Anthony Delaune
Laboratoire GPM UMR CNRS 6634 – Equipe NanoCARE Faculté des Sciences et Techniques Université de Rouen AVENUE DE L'UNIVERSITE CS 70012 76801 ST ET. DU ROUVRAY CEDEX France
ou anthony.delaune@univ-rouen.fr





